| 所属 | ① 東京大学 大学院新領域創成科学研究科 | |
|---|---|---|
| 氏名 | ① 三浦 史仁 | |
| AMED 事業 |
課題名 | メチロームおよび多重エピゲノム解析の支援 |
| 代表機関 | 東京大学 | |
| 代表者 | 三浦 史仁 | |
DNAメチル化、エピゲノム、ホルマリン固定パラフィン包埋切片
東京大学大学院新領域創成科学研究科の三浦グループは、メチローム計測の支援を行う。独自開発してきたPBAT(Post-bisulfite adaptor tagging)法を用いた全ゲノムバイサルファイトシークエンシング(Whole-genome bisulfite sequencing:WGBS)のためのライブラリー調製、次世代シークエンサーを用いた配列決定、参照ゲノム配列へのリードのマッピングと各シトシンのメチル化状態の数値化を基本的な支援として提供する。また、サンプル間でメチル化状態の異なるゲノム領域の抽出や、特定のゲノム領域毎のメチル化率の算出なども依頼者の要望に応じて支援する。
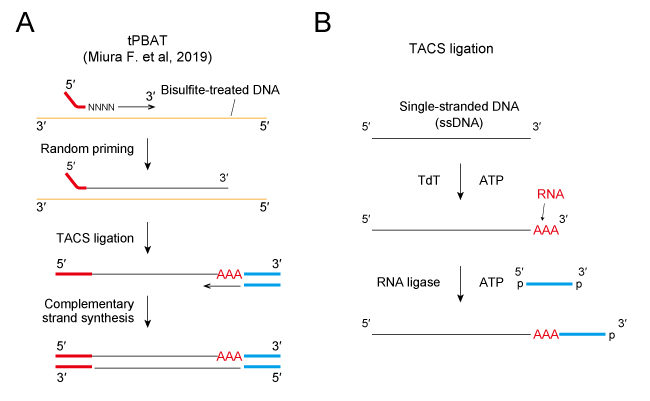
PBAT法では、数ナノグラムのゲノムDNAを開始材料としてライブラリー調製を行うことが可能である。必要なDNAの量や精製状態については、コンサルティングの上で詳細を決定するが、さまざまな量や状態のサンプルに対応してきた実績があるため、解析が困難に思えるサンプルについても、まずは相談していただきたい。
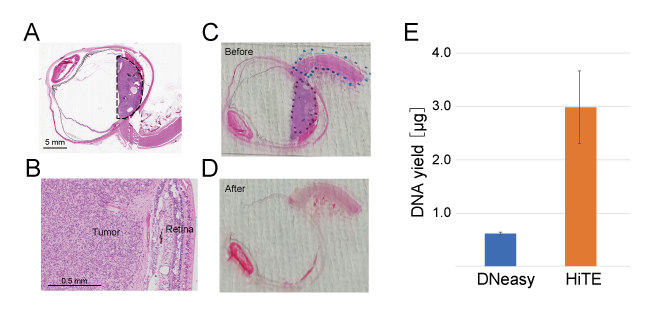
また、当グループはホルマリン固定パラフィン包埋(FFPE)切片から、高品質なDNAを高効率に回収することが可能なHiTE(Highly concentrated Tris-mediated DNA extraction)法の開発に成功した。この技術を用いることで、これまでに適用が困難だったFFPE切片に対してもWGBSを実施することが可能になった。HiTE法とPBAT法を組み合わせることにより、厚さ10 μm程度のスライスであれば、25 mm2程度の組織からWGBSによるメチローム解析を実施することが可能である。
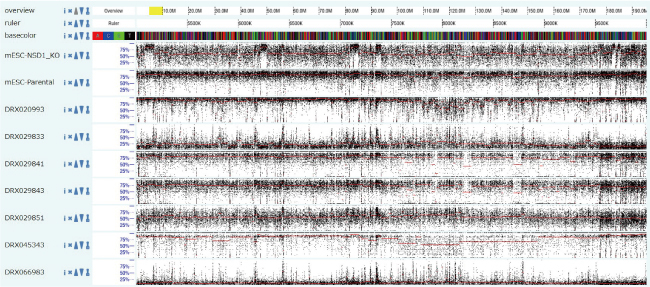
当グループは、これまで培ってきたメチローム解析技術を基盤として、多重エピゲノム解析の実現を目指したさまざまな開発を進めている。そういった技術の1つとして特定の短い塩基配列を特異的に認識してメチル化するMTaseの開発がある。例えば、CCジヌクレオチドを認識するCCMTはエピゲノム解析のプローブとして有望である。このほかにも複数のMTaseを開発して組み合わせることにより、1つのDNA分子上に複数のエピゲノム情報を書き込む多重エピゲノム解析を実現することが可能となるだろう。このような多重エピゲノム計測技術を組織切片中の特定の細胞集団に適用して、そのエピジェネティックな特性を明らかにしていくことが、このグループが目指す技術開発のゴールである。