| 所属 |
① 横浜市立大学 大学院生命医科学研究科 ② 横浜市立大学 大学院生命医科学研究科 ③ 大阪公立大学 大学院理学研究科 |
|
|---|---|---|
| 氏名 |
① 池口 満徳 ② 浴本 亨 ③ 森次 圭 |
|
| AMED 事業 |
課題名 | 構造生物学データを活用しAIと連携した分子動力学シミュレーション研究 |
| 代表機関 | 横浜市立大学 | |
| 代表者 | 池口 満徳 | |
分子動力学シミュレーション、インシリコスクリーニング、拡張アンサンブル、人工知能、スーパーコンピュータ
横浜市立大学・大阪公立大学では、タンパク質等の立体構造の分子動力学(MD)シミュレーション研究の支援を行っている。
X線結晶構造解析やクライオ電子顕微鏡によって決定された立体構造は、平均構造もしくはスナップショット構造に対応し、機能発現時、タンパク質はダイナミックに動いていると考えられている。MDシミュレーションは、そのような構造ダイナミクスを理論的に計算する研究手法であり、機能に関わる様々な情報を引き出すことが可能である。
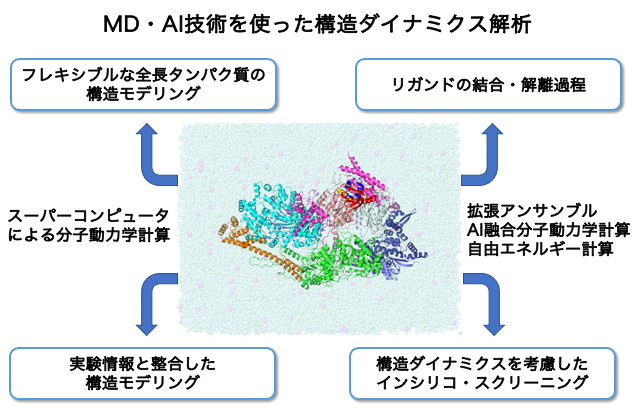
実験構造がある場合は実験構造をMDシミュレーションの初期構造として用いるが、構造が実験的に得られていない場合でも、構造モデリング、ドッキング計算やAlphaFold2などのAIによる構造予測を実施し、MDシミュレーションを実施する。また、スーパーコンピュータを用いた通常MDシミュレーションの他、拡張アンサンブルや自由エネルギー計算、AIと連携した方法など、アドバンストなMDシミュレーションも実施する。
また、インシリコスクリーニングについて、構造ダイナミクスまで考慮するため、MDシミュレーションを導入した支援を実施する。通常の分子動力学シミュレーションと結合自由エネルギー計算も併用し、インシリコスクリーニングの精度向上を図る。また、実験構造が得られていない場合でも、構造ダイナミクス解析と同様に、AlphaFold2などのAIによる構造予測を実施する。
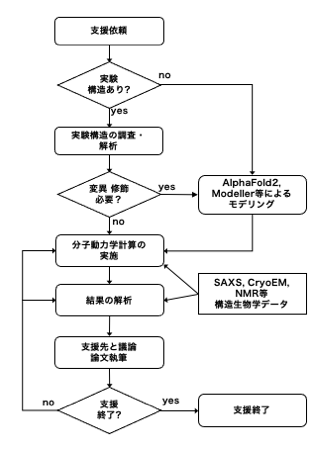
支援研究では、典型的には、下記のようなフローチャートで、支援を実施する。まず、支援依頼を受けて、構造モデリングを行う必要がある場合には、AlphaFold2, Modellerなどを用いて構造モデリングを実施する。次に、分子動力学計算を実施し、実験データと対照させながら、計算結果を解析する。その検討結果を支援先と議論して、論文執筆につなげる。
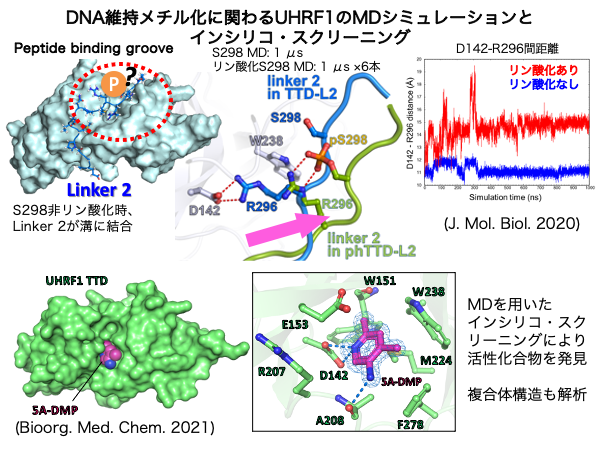
これまで行ってきた支援研究の例は下記の通りである。
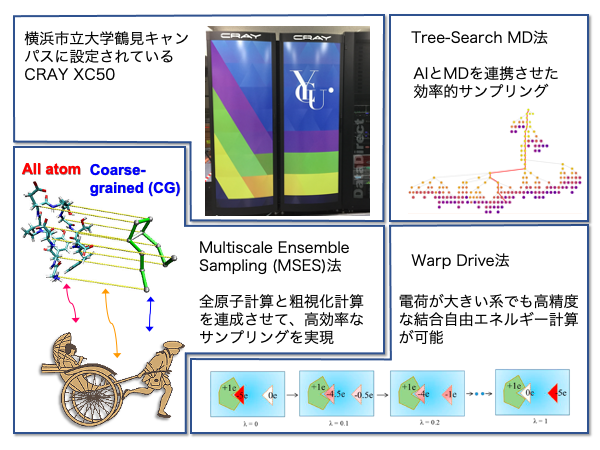
横浜市立大学と大阪公立大学からなるグループでは、分子動力学シミュレーションを軸として、研究を行っている。構造生物学等の実験研究者との共同研究が多く、X線結晶構造解析やクライオ電子顕微鏡で構造決定したタンパク質立体構造をベースにして、スーパーコンピュータ等を用いた分子動力学シミュレーションを行うことで、タンパク質のダイナミクスを解析している(上記、支援研究例を参照)。また、インシリコ・スクリーニングにおいても、分子動力学シミュレーションを活用した方法を実施している。一方、通常の分子動力学シミュレーションでは、マイクロ秒オーダーの計算が限界という問題がある。その問題を打破するための方法開発も行っている。Multiscale Ensemble Sampling (MSES)法では、全原子モデルと粗視化モデルを連成させ、高効率にタンパク質の構造サンプリングの実施が可能である。また、人工知能技術と分子動力学シミュレーションを連携させて、高効率に構造変化パスウェイを計算するTree Search (TS) MD法を開発している。さらに、電荷が大きい系でも分子間結合能を高精度に計算可能なWarp Drive法を開発している。