| 所属 |
① 早稲田大学 理工学術院 先進理工学研究科 ② 早稲田大学 理工学術院 先進理工学研究科 |
|
|---|---|---|
| 氏名 |
① 由良 敬 ② 竹山 春子 |
|
| AMED 事業 |
課題名 | 1細胞/微小組織マルチオミックスのオールインワン解析による生命科学研究の支援 |
| 代表機関 | 早稲田大学 | |
| 代表者 | 由良 敬 | |
トランスクリプトーム解析、メタボロミクス、プロテオミクス、データベース、分子動力学シミュレーション
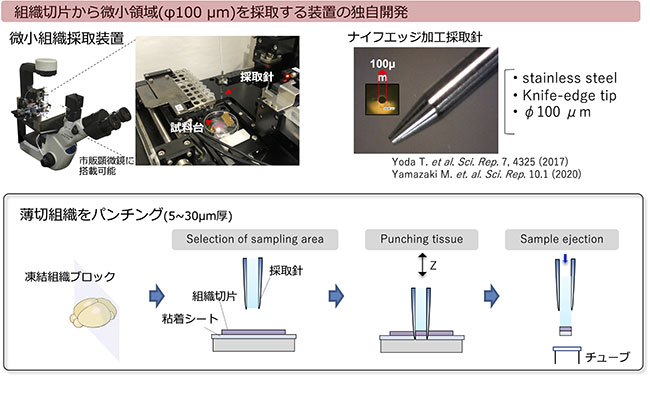
この支援では、1細胞/微小組織(空間的位置情報付)からはじまるDNA/RNA解析、プロテオーム解析、メタボローム解析、およびバイオインフォマティクス解析のオールインワン解析を実施する。従来の支援で問題となっていたウェットとドライの分離を回避するために、発現・機能解析ユニットとインシリコ解析ユニットが連携し、研究支援コンサルティングの段階からウェットとドライが融合して支援課題に取り組む。1細胞RNA解析と、独自の微小組織採取装置(Yoda et al, Sci rep, 2017)を活用した空間的位置情報を担保した微小組織DNA/RNA-seqライブラリ調製法(Yamazaki et al, Sci rep, 2020)を利用する。この技術により創薬に直結する臨床検体の解析で必要とされる、貴重な試料から多角的な分析や対象部位を絞った分析を実現する。さらにプロテオームやメタボローム情報も細胞機能を理解する上では重要であり、連携ユニット分担機関の九州大学がもつ世界最高精度の各種クロマトグラフィーおよび質量分析技術を用いて、微小組織サンプルに含まれる代謝物およびタンパク質の超網羅的かつ定量的な情報が取得できる(Hata et al, Anal Chem, 2020; Nakatani et al, Mass Spectrum, 2020)。mRNAは全長解析が可能であり、細胞ごとの選択的スプライシングの違いを解析することもできる。ここで得られるデータを、早稲田大学と東京大学でインシリコ解析する。ノンコーディングRNAを含むRNA情報、発現タンパク質、タンパク質と相互作用するメタボライトの情報を取得する。また、一般的なタンパク質解析ツールに加えて、高精度アラインメントツールや発現タンパク質のオーソログ解析ツール、ホモロジーモデリングツール、低分子ドッキングツール、RNA解析ツールとしてRNAの二次構造予測ツールやRNAの多重アラインメントツール、 RNA-RNA相互作用予測ツール、構造予測統合Webサーバなどによる解析を支援実施する。東京大学では文献からの関連情報抽出を実施できる(Muramatsu et al, Bioinfo. Adv., 2021)。これらの技術開発経験および解析経験を活かした支援を行う。これらの解析結果を支援申請者に提供する際には、データの解釈を協議する研究支援内容ディスカッションテーブルを開催する。このテーブルにおいては、支援申請者に対して解析結果を丁寧に説明するともに、論文執筆や特許取得に関するディスカッションも行う。
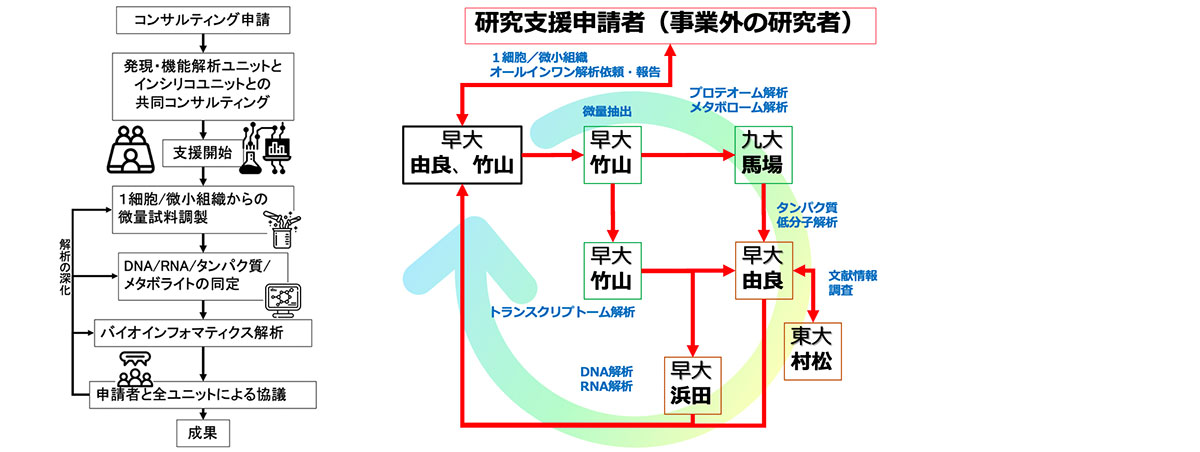
この支援では、以下の技術を統合して適用する。
【トランスクリプトーム解析支援例】
【メタボローム・プロテオーム解析支援例】

【トランスクリプトーム・インシリコ解析支援例】
【データベース/モデリング解析支援例】
【分子動力学シミュレーション解析支援例】
【文献情報解析支援例】
【トランスクリプトーム解析支援(早稲田大学竹山グループ)】
微小組織採取システムにより、顕微鏡画像上でユーザーが選択した領域を直径100ミクロンのサイズで高速・自動でサンプリングすることができる。採取した微小組織片のRNA-seq解析と、組織の画像情報を組み合わせることで、組織内の遺伝子発現を微小空間の解像度で捉える。組織採取の効率化を目指したシステム高度化、mRNA発現情報以外の情報も含めたマルチオミクス解析手法の開発、組織画像と遺伝子発現の統合解析プラットフォームの構築を推進する。また、一粒子レベルでのウイルスゲノム解析技術の開発を進め、ホストと病原体(ウイルス)変異との感染応答の双方向解析の支援提供を目指す。
【メタボローム・プロテオーム解析支援(九州大学馬場グループ)】
プロテオミクスおよびメタボロミクスは質量分析(MS: mass spectrometry)の技術革新の恩恵を受け発展してきた学問領域である。タンパク質および代謝物は、生命システムを理解するための重要分子であると同時にゲノム情報の実行の結果、すなわち高解像度のフェノタイプとしても捉えることができる。しかし、生体内に存在する膨大かつ多様な代謝物およびタンパク質の分子種を網羅的かつ定量的に計測することは未だ簡単ではない。我々は、これまで各種クロマトグラフィー高分解能タンデム質量分析を基盤とした世界最高感度かつ網羅的なメタボロームおよびプロテオーム分析法を開発してきた。2022年度は、体液(血漿50 μL程度)、細胞(1×106個程度)、組織(10 mg FW程度)などの生体試料を対象としたメタボローム解析およびプロテオーム解析を行い、代謝制御のメカニズム解明や新規のバイオマーカーの発見などに貢献できる研究支援を実施する。
【トランスクリプトーム・インシリコ解析支援(早稲田大学浜田グループ)】
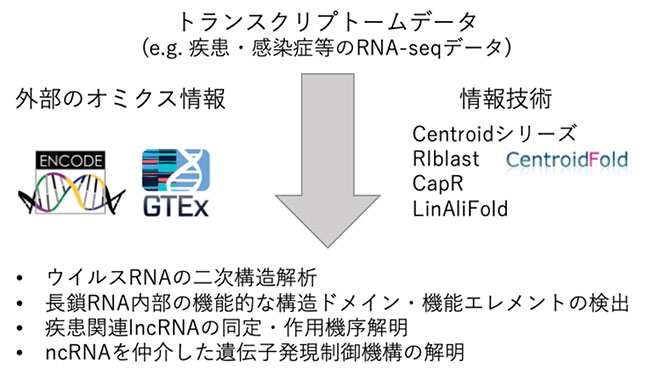
これまでに、RNA二次構造解析を基盤とした、多様なRNA情報解析技術を開発してきた。たとえば、世界最高クラスの予測精度を持つCentroidFold [Bioinformatics. 2009 Feb 15;25(4):465-73]や、トランスクリプトームレベルでのRNA-RNA相互作用を予測可能とする世界最速の相互予測ソフトウェアRIblast [Bioinformatics. 2017 Sep 1;33(17):2666-2674]、ウイルス配列にも適用可能な高速高精度構造予測手法のLinAliFold [論文準備中] などがある。また、ノンコーディングRNA特に長鎖ノンコーディングRNAの機能の解明に向けたオミクスデータを活用した研究を行っている [Mob DNA. 2021 Jan 20;12(1):3; NAR Genom Bioinform. 2021 Jul 2;3(3):lqab055; BMC Genomics. 2018 Dec 31;19(Suppl 10):906など]。
本課題では、これまで開発を行ってきた技術について、精度・速度の面でさらなる高度化をはかるとともに、シュードノット構造を考慮できるようにするなどの機能拡充を図る。また、これらのソフトウェアを活用して、疾患関連遺伝子など特定のタンパク質やncRNAを対象として、それらを制御する発現制御機構を明らかにする研究を行う。
【タンパク質の機能推定解析支援(早稲田大学由良グループ)】
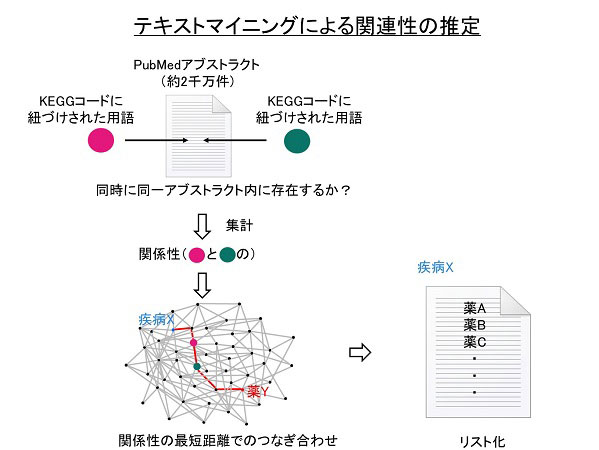
【文献情報解析支援(東京大学村松グループ)】
テキストマイニングによる関連性推定法の開発とその新型コロナウイルス関連薬推定への応用については論文として公表した(Muramatsu, T. & Tanokura, M. Bioinformatics Advances 1(1) vbab013 (2021))。構造生物学的、酵素学的研究も行っており、コロナウイルス主要プロテアーゼの自己プロセシング過程の解析、その際の特殊な特異異性発現とその機構を明らかにしてきた(Muramatsu et al. Proc. Natl. Acad. Sci. USA, 113, 12997-13002 (2016); Muramatsu et al. FEBS J. 280, 2002-2013 (2013))。
その他のタンパク質構造機能相関研究、食品等の分析なども行っている。